概要
DScribeは、材料科学向け機械学習で用いる原子環境記述子を高速に生成できるPythonパッケージである。本レビューでは、Cu(111)表面の吸着サイト解析と小分子の構造類似度評価を通じて、DScribeの特徴・使い勝手・応用可能性を検証した。
DScribeとは?
DScribeは、SOAP・MBTR・Coulomb matrixなど複数の原子記述子を提供し、ASEやscikit-learnと連携しながら材料データの特徴抽出を支援するライブラリである。プロジェクトはAalto UniversityのSINGROUPが開発しており、Apache License 2.0の下で公開されている。
主要な特徴
- SOAP/MBTR/Coulomb matrixなど多様な記述子に対応
- ASEの
AtomsオブジェクトやNumPy配列とシームレスに連携 - 並列計算やバッチ処理に対応し、1万点規模の記述子計算も実用的な時間で完了
- scikit-learn互換のカーネル(AverageKernel, REMatchKernel)を備え、類似度評価やMLパイプラインに直接組み込める
インストール
pipから容易に導入できる。依存パッケージとしてsparseやnumbaが自動的に追加される。
python3 -m pip install --user dscribe ase scikit-learn matplotlib
使用例1: Cu(111)吸着サイトのクラスタリング
公式ドキュメントでは、表面から1 Å上方に設定したSOAP記述子の格子をk-meansでクラスタリングし、吸着候補を代表点に絞り込む手順が紹介されている。チュートリアル同様に10クラスタへ分類することで、より高価な計算や教師あり学習に投入する点数を効率的に削減できると説明されている。本レビューでも同じレシピに従い、Cu(111)表面上の1万点に対してMiniBatchKMeansを適用した。数秒でクラスタリングが完了し、対称等価な吸着位置ごとにグルーピングされる。
import numpy as np
import ase.build
from dscribe.descriptors import SOAP
from sklearn.cluster import MiniBatchKMeans
# Cu(111)表面の構築
system = ase.build.fcc111("Cu", (4, 4, 4), a=3.597, vacuum=10.0, periodic=True)
# SOAP記述子
soap = SOAP(
species=["Cu"],
r_cut=5.5,
sigma=0.1,
n_max=12,
l_max=12,
periodic=True,
weighting={"function": "poly", "r0": 12, "m": 2, "c": 1, "d": 1},
)
# 100×100の格子を走査
cell = system.get_cell()
top = (system.get_positions()[:, 2].max() + 1.0) / cell[2, 2]
grid = np.linspace(0.0, 1.0, 100)
x, y, z = np.meshgrid(grid, grid, [top])
positions = cell.cartesian_positions(np.vstack([x.ravel(), y.ravel(), z.ravel()]).T)
D = soap.create(system, positions)
# MiniBatchKMeansによるクラスタリング
model = MiniBatchKMeans(n_clusters=10, batch_size=1024, random_state=42, n_init=5)
labels = model.fit_predict(D)
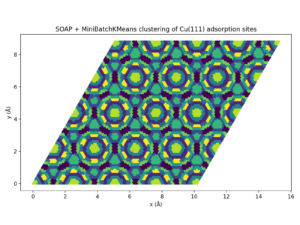
SOAP記述子とMiniBatchKMeansによるCu(111)吸着サイトのクラスタリング結果”
使用例2: 小分子の構造類似度評価
「Similarity Analysis」チュートリアルでは、局所環境ベクトルだけでなく「平均カーネルで全原子の寄与を足し合わせる方法」と「REMatchカーネルで最適マッチングと平均化をαパラメータで補間する方法」が提案されている。チュートリアル本編ではH2OとH2O2を例に挙げ、REMatchを用いる際には事前正規化が必須である点が強調されている。本レビューではその流れを拡張し、水・過酸化水素・アンモニア・メタンの4分子で両手法を比較した。正規化したSOAP記述子から線形・RBF双方の類似度行列を取得し、ヒートマップとして可視化している。
from dscribe.descriptors import SOAP from dscribe.kernels import AverageKernel, REMatchKernel from ase.build import molecule from sklearn.preprocessing import normalize names = ["H2O", "H2O2", "NH3", "CH4"] structures = [molecule(name) for name in names] soap = SOAP(species=["H", "C", "N", "O"], r_cut=5.0, sigma=0.2, n_max=2, l_max=2, periodic=False) features = [soap.create(struct) for struct in structures] features_norm = [normalize(f) for f in features] avg_kernel = AverageKernel(metric="linear").create(features) re_kernel = REMatchKernel(metric="rbf", gamma=1.0, alpha=1.0).create(features_norm)

応用分野
- 触媒・表面科学: 吸着サイトのクラスタリング、活性点の選定
- 固体材料探索: SOAPやMBTRを用いた類似構造検索、データ駆動型スクリーニング
- 分子設計: REMatchカーネルを使った分子間の定量的な類似度評価
- 機械学習モデル構築: 生成した記述子を回帰・分類・クラスタリングに投入し、活性や安定性を予測
類似ツールとの比較
- ASEのみ: ASEには記述子生成機能が限られるため、特徴抽出には追加実装が必要。DScribeは豊富な記述子を標準で提供。
- quippy/libatoms: GAPポテンシャル用の記述子生成が可能だが、Pythonでの汎用利用やドキュメント整備ではDScribeが扱いやすい。
- Matminer: 材料データベース向けの特徴量が豊富だが、局所環境ベースのSOAPなどはDScribeの方が高速・柔軟。
使ってみた感想
- 操作性: ASE・NumPyと同じ感覚で扱え、公式サンプルをほぼそのまま再現できました。
- 計算速度: 1万点×1014次元のSOAP計算でも約6秒で完了(Apple Silicon M4 Pro)。ノートPCでも十分実用的。
- 安定性:
MiniBatchKMeansはrandomstateとninitを指定することで再現性が確保でき、REMatchKernelはsklearn.preprocessing.normalizeとの併用で数値的に安定。 - ワークフロー: 記述子ベクトル(
D.npy)と座標(r.npy)を保存すれば、可視化・最適化・機械学習モデルに容易に引き継げる。
結論
DScribeは、材料科学における機械学習ワークフローを加速する強力なツールである。表面吸着解析や分子類似度評価など幅広い応用に対応し、既存のPythonエコシステムと統合しやすい点が魅力であった。MatDaCs掲載ツールとして、記述子生成・類似度解析を試したい研究者に強く推奨できる。
参考資料
- DScribeドキュメント: <https://singroup.github.io/dscribe/latest/>
- チュートリアル(クラスタリング): <https://singroup.github.io/dscribe/latest/tutorials/machine_learning/clustering.html>
- チュートリアル(Similarity Analysis): <https://singroup.github.io/dscribe/latest/tutorials/similarity_analysis/kernels.html>
- GitHubリポジトリ: <https://github.com/SINGROUP/dscribe>

